In diesem Leitfaden werden wir zunächst den Ansatz sehen und dann den Schlaganfall jeder Person vorhersagen, indem wir die Daten Schritt für Schritt analysieren.
Logistische Regression
Die logistische Regression, auch „Logit-Modell“ genannt, wird in der prädiktiven Analyse für Klassifizierungsprobleme verwendet. Dies ist ein Modell des maschinellen Lernens, das nur mit der Klassifizierung arbeitet, um die Klassen (Kategorien) zu klassifizieren. Beispielsweise gibt es in unserem Szenario zwei Kategorien (eine Person, die von einem Schlaganfall betroffen ist, und eine Person, die nicht von einem Schlaganfall betroffen ist). Die besten Anwendungen des Modells sind die Vorhersage von Herzerkrankungen, die Vorhersage des Geschlechts, die Vorhersage der Pflanzenlebensdauer usw.
Schritte:
1. Datenerfassung: Die Daten sind für die Durchführung von Vorhersagen/Analysen erforderlich. Es kann unter anderem im Format CSV/XLSX vorliegen. Wir können dies mit der Methode spark.read.csv() in die Spark-Umgebung (DataFrame) laden.
2. Datenanalyse : Die Analyse der Attribute/Spalten wird als „Datenanalyse“ bezeichnet. Die Spalten, die zur Vorhersage der Klasse beitragen, werden als „unabhängige Attribute“ bezeichnet. Die Spalte, die zur Vorhersage führt, wird als „abhängiges oder Zielattribut“ bezeichnet. In diesem Szenario können wir die Eigenschaft columns verwenden, um alle Spalten anzuzeigen. Die Methode „distinct()“ wird verwendet, um die eindeutigen Werte anzuzeigen.
3. Datenvorverarbeitung: Das Filtern der Null-/fehlenden Werte wird als „Vorverarbeitung“ bezeichnet. In dieser Phase entfernen wir alle fehlenden Werte. Die Maschine kennt nur die Binärsprache. Daher sollten alle Zeichenfolgenkategorien in numerische kategoriale Werte konvertiert werden. In PySpark können wir „StringIndexer“, die verfügbare Klasse im Modul pyspark.ml.feature, verwenden, um die Zeichenfolgenkategorien in numerische Kategorien umzuwandeln. Es konvertiert sie automatisch intern. Wir müssen die Werte nicht angeben. Sie können die folgende Syntax verwenden:
indexer_data=StringIndexer(inputCol= „String_Category_ColumnName“ ,outputCol= 'New_Column_name' )
4. Vektormontage: Jetzt haben Sie die Daten, die die Maschine verstehen kann. In dieser Phase sollten alle unabhängigen Attribute in einer einzigen Spalte vektorisiert werden. Dies kann mit der VectorAssembler-Klasse erfolgen. Es benötigt zwei Parameter: Der erste Parameter ist inputCols, der eine Liste unabhängiger Attribute akzeptiert. Der zweite Parameter ist die Ausgabespalte, die alle Eingabespalten in diese Variable vektorisiert.
assembler=VectorAssembler(inputCols=[Spalten…],outputCol=vectorized_data)5. Transformation: Bereiten Sie nun Ihre Daten vor, indem Sie die aktualisierten Spalten (Schritt 3) mit der Funktion transform() transformieren.
assembler.transform(inxed_data)
6. Vorbereiten der Daten für Training und Tests: In dieser Phase teilen wir die Daten in „Training“ und „Testen“ auf. Es ist besser, wenn wir die 70 % der Daten zum Trainieren des Modells und die 30 % der Daten zum Testen des Modells aufteilen. Dies kann mit der Methode randomSplit() erreicht werden. Es benötigt eine Liste, die zwei Float-Werte enthält: einen für die Testaufteilung und einen für die Zugaufteilung.
train_data,test_data=final_data.select([ 'Merkmale' ,target_column]).randomSplit([0.70.0.30])7. Modellanpassung und -bewertung : Es ist Zeit, das logistische Regressionsmodell anzupassen. Das logistische Regressionsmodell ist im Modul pyspark.ml.classification verfügbar. Es übernimmt die Klassenbezeichnung/Zielspalte. Dies führt zu den Spalten „rawPrediction“, „Wahrscheinlichkeit“ und „Prediction“. Unsere Ergebnisse werden in der Vorhersagespalte gespeichert.
# Modellanpassunglogistic_regression_model=LogisticRegression(labelCol=target_column).fit(train_data)
# Modellbewertung
train_results=logistic_regression_model.evaluate(train_data).predictions
8. Genauigkeit und Ergebnisse: Dies ist die letzte Phase, in der wir die Modellgenauigkeit mithilfe beliebiger Testtechniken testen.
Schauen wir uns das folgende Projekt an, in dem wir mithilfe des logistischen Regressionsmodells vorhersagen, ob eine Person von einem Schlaganfall betroffen ist oder nicht.
Schlaganfallvorhersage
9. Installieren Sie das PySpark-Modul in Ihrer Umgebung. Folgendes ist der zu installierende Befehl:
Pip Pyspark installieren 
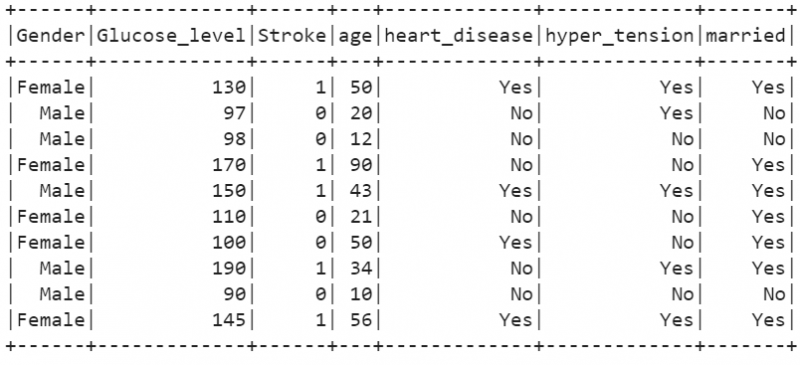
10. Erstellen wir einen PySpark DataFrame mit 10 Datensätzen, die sich auf Schlaganfalldetails von 10 Personen beziehen. Zur Demonstration erstellen wir einen DataFrame ohne CSV. Dieser DataFrame enthält 7 Spalten. „Geschlecht“, „Glucose_level“, „married“, „age“, „heart_disease“, „hyper_tension“ sind unabhängige Attribute und „Stroke“ ist die Klassenbezeichnung oder das abhängige Attribut. Das bedeutet, dass der Schlaganfall eines Menschen von diesen unabhängigen Eigenschaften abhängt.
Pyspark importierenaus pyspark.sql SparkSession importieren
linuxhint_spark_app = SparkSession.builder.appName( „Linux-Hinweis“ ).getOrCreate()
stoke_data =[
{ 'Geschlecht' : 'Weiblich' , 'Alter' : fünfzig , 'Herzkrankheit' : 'Ja' ,
'Hypertonie' : 'Ja' , 'verheiratet' : 'Ja' , 'Glucose_level' : 130 , 'Schlaganfall' : 1 },
{ 'Geschlecht' : 'Männlich' , 'Alter' : zwanzig , 'Herzkrankheit' : 'NEIN' ,
'Hypertonie' : 'Ja' , 'verheiratet' : 'NEIN' , 'Glucose_level' : 97 , 'Schlaganfall' : 0 },
{ 'Geschlecht' : 'Männlich' , 'Alter' : 12 , 'Herzkrankheit' : 'NEIN' ,
'Hypertonie' : 'NEIN' , 'verheiratet' : 'NEIN' , 'Glucose_level' : 98 , 'Schlaganfall' : 0 },
{ 'Geschlecht' : 'Weiblich' , 'Alter' : 90 , 'Herzkrankheit' : 'NEIN' ,
'Hypertonie' : 'NEIN' , 'verheiratet' : 'Ja' , 'Glucose_level' : 170 , 'Schlaganfall' : 1 },
{ 'Geschlecht' : 'Männlich' , 'Alter' : 43 , 'Herzkrankheit' : 'Ja' ,
'Hypertonie' : 'Ja' , 'verheiratet' : 'Ja' , 'Glucose_level' : 150 , 'Schlaganfall' : 1 },
{ 'Geschlecht' : 'Weiblich' , 'Alter' : einundzwanzig , 'Herzkrankheit' : 'NEIN' ,
'Hypertonie' : 'NEIN' , 'verheiratet' : 'Ja' , 'Glucose_level' : 110 , 'Schlaganfall' : 0 },
{ 'Geschlecht' : 'Weiblich' , 'Alter' : fünfzig , 'Herzkrankheit' : 'Ja' ,
'Hypertonie' : 'NEIN' , 'verheiratet' : 'Ja' , 'Glucose_level' : 100 , 'Schlaganfall' : 0 },
{ 'Geschlecht' : 'Männlich' , 'Alter' : 3. 4 , 'Herzkrankheit' : 'NEIN' ,
'Hypertonie' : 'Ja' , 'verheiratet' : 'Ja' , 'Glucose_level' : 190 , 'Schlaganfall' : 1 },
{ 'Geschlecht' : 'Männlich' , 'Alter' : 10 , 'Herzkrankheit' : 'NEIN' ,
'Hypertonie' : 'NEIN' , 'verheiratet' : 'NEIN' , 'Glucose_level' : 90 , 'Schlaganfall' : 0 },
{ 'Geschlecht' : 'Weiblich' , 'Alter' : 56 , 'Herzkrankheit' : 'Ja' ,
'Hypertonie' : 'Ja' , 'verheiratet' : 'Ja' , 'Glucose_level' : 145 , 'Schlaganfall' : 1 }
]
# Erstellen Sie den Datenrahmen aus den oben genannten Daten
Stroke_df = linuxhint_spark_app.createDataFrame(stoke_data)
# Tatsächlicher stoke_df
Stroke_df.show()
Ausgang:
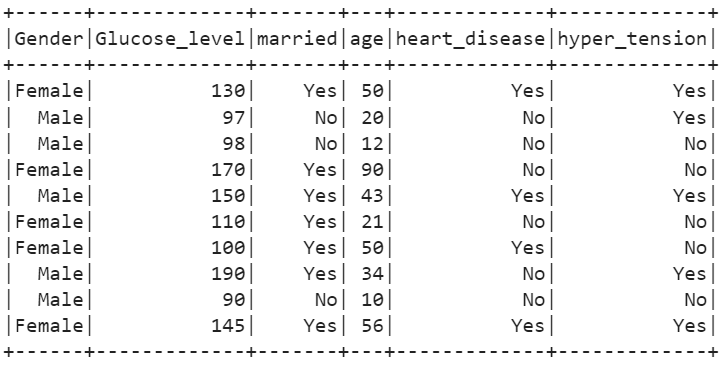
11. Zeigen Sie die unabhängigen Spalten mit der Methode select() an.
# Unabhängige Attribute anzeigenStroke_df.select(Stroke_df[ 'Geschlecht' ],Stroke_df[ „Glucose_level“ ],Stroke_df[ 'verheiratet' ],Stroke_df[ 'Alter' ],Stroke_df[ 'Herzkrankheit' ],Stroke_df[ 'Hypertonie' ]).zeigen()
Ausgang:
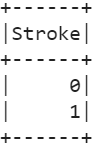
12. Zeigen Sie die eindeutigen Werte an, die im Zielattribut (Stroke) vorhanden sind.
# Eindeutige Werte des ZielattributsStroke_df.select(Stroke_df[ 'Schlaganfall' ]).distinct().show()
Ausgang:
13. Geben Sie den Datentyp aller Spalten mit der Funktion printSchema() zurück.
# Gibt den Datentyp aller Spalten zurück.Stroke_df.printSchema()
Ausgang:

Wir können sehen, dass 4 Spalten vom Typ String sind. Lassen Sie uns sie in kategoriale numerische Werte umwandeln.
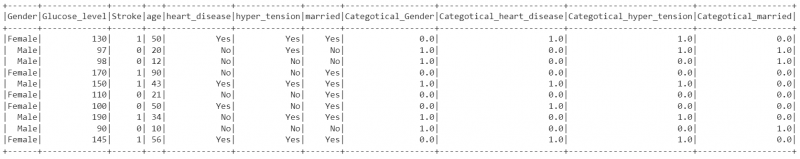
14. Konvertieren wir die kategorialen String-Werte mithilfe des StringIndexer in den Spalten „Gender“, „heart_disease“, „hyper_tension“ und „married“ in numerische kategoriale Werte und schreiben diese in die Spalten Categotical_Gender, Categotical_heart_disease, Categotical_hyper_tension und Categotical_married. Speichern Sie die Spalten mit der Methode fit() im DataFrame indexed_data.
aus pyspark.ml.feature StringIndexer importieren# Konvertieren Sie kategoriale String-Werte in numerische kategoriale Werte in der Spalte „Geschlecht“.
indexer_data=StringIndexer(inputCol= 'Geschlecht' ,outputCol= 'Categotical_Gender' )
indexed_data=indexer_data.fit(Stroke_df).transform(Stroke_df)
# Konvertieren Sie kategoriale String-Werte in numerische kategoriale Werte in der Spalte „heart_disease“.
indexer_data=StringIndexer(inputCol= 'Herzkrankheit' ,outputCol= „Categotical_heart_disease“ )
indexed_data=indexer_data.fit(indexed_data).transform(indexed_data)
# Konvertieren Sie kategoriale String-Werte in numerische kategoriale Werte in der Spalte „hyper_tension“.
indexer_data=StringIndexer(inputCol= 'Hypertonie' ,outputCol= 'Categotical_hyper_tension' )
indexed_data=indexer_data.fit(indexed_data).transform(indexed_data)
# Konvertieren Sie kategoriale Zeichenfolgenwerte in numerische kategoriale Werte in der Spalte „verheiratet“.
indexer_data=StringIndexer(inputCol= 'verheiratet' ,outputCol= 'Categotical_married' )
indexed_data=indexer_data.fit(indexed_data).transform(indexed_data)
# Zeigt die aktualisierte Version an
indexed_data.show()
Ausgang:
15. Konvertieren Sie die unabhängigen Spalten mit dem Vektorassembler in einen Vektor. Der Vektorname lautet „features“.
aus pyspark.ml.feature VectorAssembler importierenassembler= VectorAssembler(inputCols=[ 'Categotical_Gender' , „Categotical_heart_disease“ , 'Categotical_hyper_tension' , 'Categotical_married' , 'Alter' ,
'Glucose_level' ],outputCol= 'Merkmale' )
16. Transformieren Sie die vorherigen Daten mit der Funktion transform() in einen endgültigen DataFrame und zeigen Sie ihn mit der Funktion show() an.
Ausgang:

17. Bereiten Sie die Daten für Training und Tests vor, indem Sie sie in 70–30 aufteilen. Die „Features“ werden in den train_data und „Stroke“ in den test_data gespeichert.
# Bereiten Sie Daten für Schulung und Tests vortrain_data,test_data=final.select([ 'Merkmale' , 'Schlaganfall' ]).randomSplit([ 0,70 , 0,30 ])
18. Passen Sie das logistische Regressionsmodell an und bewerten Sie es.
aus pyspark.ml.classification Import LogisticRegression# Modellanpassung
logistic_regression_model=LogisticRegression(labelCol= 'Schlaganfall' ).fit(train_data)
# Modellbewertung
train_results=logistic_regression_model.evaluate(train_data).predictions
train_results.show()
Ausgang:
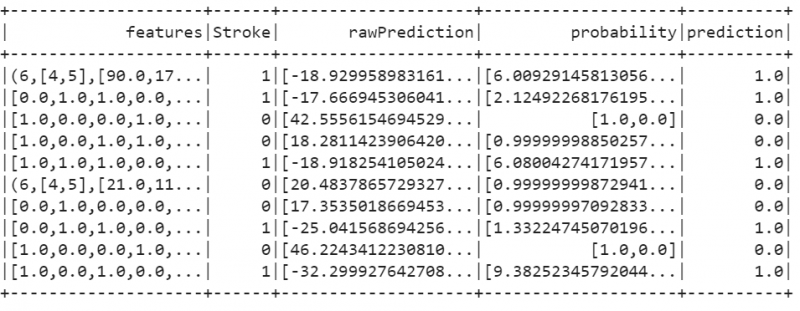
19. Verwenden Sie den BinaryClassificationEvaluator, um die Modellgenauigkeit zu überprüfen. Wir können sehen, dass unser Modell 100 % genau ist.
aus pyspark.ml.evaluation importieren BinaryClassificationEvaluator# Rufen Sie den BinaryClassificationEvaluator auf
results = BinaryClassificationEvaluator(rawPredictionCol= 'Vorhersage' ,labelCol= 'Schlaganfall' )
ROC_AUC = results.evaluate(train_results)
print(ROC_AUC * 100 , '% Genauigkeit' )
Abschluss
Mithilfe des logistischen Regressionsmodells in PySpark können Sie das Problem der Klassifizierung realer Kacheln vorhersagen. Das Entfernen der Nullwerte/fehlenden Werte ist wichtig, da sie die Modellauswertung und -genauigkeit beeinträchtigen. Die Vorverarbeitung ist ein notwendiger Schritt vor der Anpassung eines maschinellen Lernmodells. Stellen Sie sicher, dass Sie diesen Schritt richtig und genau ausführen.